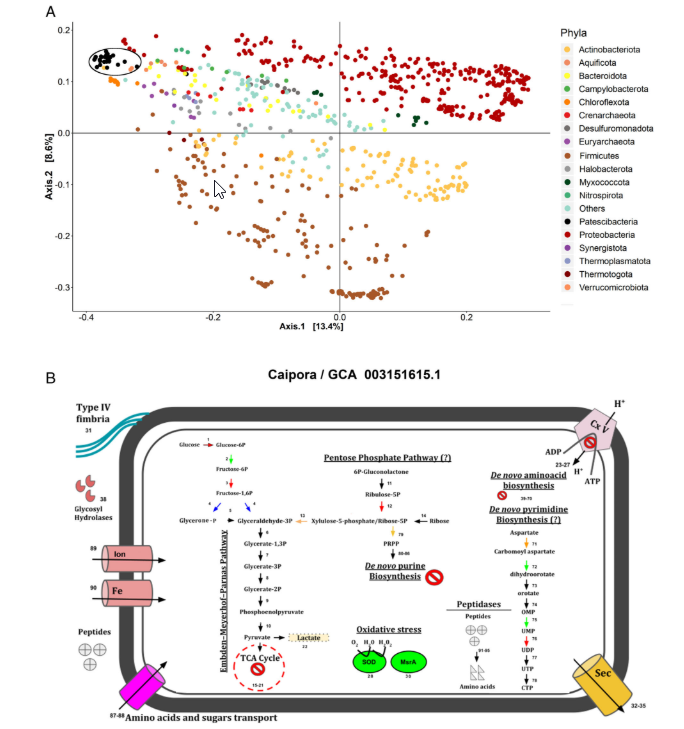
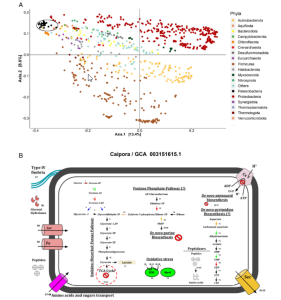
Abstract
Soil microbiome is one of the most heterogeneous biological systems. State-of-the-art molecular approaches such as those based on single-amplified genomes (SAGs) and metagenome assembled-genomes (MAGs) are now improving our capacity for disentailing soil microbial-mediated processes. Here, we analysed publicly available datasets of soil microbial genomes and MAG’s reconstructed from the Amazon’s tropical soil (primary forest and pasture) and active layer of permafrost, aiming to evaluate their genome size. Our results suggest that the Candidate Phyla Radiation (CPR)/Patescibacteria phyla have genomes with an average size fourfold smaller than the mean identified in the RefSoil database, which lacks any representative of this phylum. Also, by analysing the potential metabolism of 888 soil microbial genomes, we show that CPR/Patescibacteria representatives share similar functional profiles, but different from other microbial phyla and are frequently neglected in the soil microbial surveys. Finally, we argue that the use of MAGs may be a better choice over SAGs to expand the soil microbial databases, like RefSoil.